| Recherche en Bioinformatique - Alignement |
|
|
| Cette page est un bref aperçu des notions de base en rapport avec l'alignement de séquences en bioinformatique. |
| En quoi consiste l'alignement ? |
L'alignment de séquences d'ADN ou d'acides aminés est une opération de base en bioinformatique qui a pour but d'identifier des zones conservées entre séquences. L'alignement sert notamment à :
Dans la compréhension du fonctionnement de la vie, les protéines jouent un rôle essentiel. On part donc de l'hypothèse que des protéines comportant des séquences similaires risquent fort de posséder des propriétés physico-chimiques identiques :
| à partir de l'identification de similarités entre une première séquence dont on connait le mécanisme d'action et une deuxième séquence dont on ne connait pas le mécanisme de fonctionnement, on peut inférer des similarités structurelles ou fonctionnelles sur la séquence non connue et proposer de vérifier de manière expérimentale le comportement d'action supposé. |
ACATT } et S1 = { AAGTT }, un alignement de S1 et S2 est par exemple :| S1 : | A |
C |
A |
- |
T |
T |
| S2 : | A |
- |
A |
G |
T |
T |
| Consensus : | A |
. |
A |
. |
T |
T |
| Cadre formel |
| Définition 1 - Alphabet |
Un alphabet 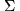 est un ensemble fini de symboles disctincts. Dans le cas de séquences d'ADN ou d'acides aminés on définit le symbole vide ou gap par est un ensemble fini de symboles disctincts. Dans le cas de séquences d'ADN ou d'acides aminés on définit le symbole vide ou gap par -.
|
L'alphabet de l'ADN est composé par les symboles :
-, A, C, G, T
L'alphabet des acides aminés est composé des symboles :
-, A, C, D, E, F, G, H, I, K, L, M, N, P, Q, R, S, T, V, W, Y
| Définition 2 - Séquence, segment |
On appelle séquence S une suite ordonnée de caractères pris dans un alphabet 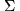 : :
|
| Définition 3 - Alignement | |||
|
Soit S = { S1, S2, ..., Sk } un ensemble de k séquences. Un alignement de S, noté A(S1, S2, ..., Sk) est une matrice :
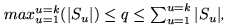
telle que |
|
Jean-Michel Richer, 2004