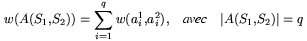La ressemblance ou similarité de deux séquences d'ADN peut être expliquée en prenant comme postulat de départ que toutes les espèces sont issues d'un même ancêtre originel. Selon cette théorie, des mutations ont eu lieu au cours de l'évolution générant des séquences qui ont donné naissance à des espèces de natures différentes. La plupart des changements sont dus à des mutations locales sur les brins d'ADN qui sont soient :
- des insertions d'un nucléotide ou d'un ensemble de nucléotides,
- des suppressions d'un nucléotide ou d'un ensemble de nucléotides,
- des substitutions d'un nucléotide par un autre nucléotide.
Deux séquences peuvent donc être proches ou éloignées. Il existe plusieurs degrés de ressemblance (ou de similarité) entre séquences :
- l'identité : deux séquences sont dites identiques si elles se correspondent parfaitement. La notion d'identité fait également référence au pourcentage de résidus identiques en regard dans un alignement,
- la similarité : deux séquences sont dites similaires si elles sont relativement proches sans toutefois être identiques,
- l'homologie est un cas particulier de similarité dans lequel on considère que deux séquences similaires dérivent d'un ancêtre commun, c'est-à-dire que les séquences sont des évolutions d'une séquence initiale commune pour laquelle certaines parties ont été ajoutées, supprimées ou remplacées.
Matrice de score
 |
L'opération d'alignement consiste à faire apparaître le possible cheminement d'une évolution d'une séquence par rapport à une autre ou des deux séquences par rapport à un hypothétique ancêtre commun.
L'opération d'alignement consistera donc à mettre en regard les caractères de deux séquences S1 et S2 de manière à ce que les séquences se correspondent le mieux possible. Les séquences n'étant de manière générale pas strictement identiques on autorise le décalage des caractères en insérant des espaces (ou gap) dans les séquences.
Un alignement peut être construit grâce à quatre opérations de base :
- l'appariement (match) : (a,a), un caractère de la première séquence est mis en regard d'un caractère identique dans la deuxième séquence,
- la substitution : (a,b), un caractère de la première séquence est mis en regard d'un caractère différent dans la deuxième séquence,
- l'insertion d'un gap dans S1 : (-,b)
- l'insertion d'un gap dans S2 : (a,-)
Remarque : l'insertion dans une séquence peut également être considérée comme une suppression (ou deletion en anglais) dans la séquence en regard. On parle alors d'opérateurs d'indel (INsertion-DELetion).
Exemple
Soient les séquences S1 = {CATTGC} et S2 = {ACAGTC}. Un exemple d'alignement est :
La construction de cet alignement correspond à la suite d'opérations suivantes :
| (-,A) | insertion d'un gap dans S1 |
| (C,C) | appariement |
| (A,A) | appariement |
| (T,G) | substitution |
| (T,T) | appariement |
| (G,-) | insertion d'un gap dans S2 |
| (C,C) | appariement |
Cependant, d'autres alignements semblent tout aussi valables :
|
Afin de déterminer le ou les meilleur(s) alignment(s), on attribue un coût à chaque opération d'alignement suivant une matrice de score (notée w) et on tente d'optimiser le coût de l'ensemble de ces opérations. Un exemple simple de matrice de score est la matrice identité :