CUDA : cours travaux dirigés
Sommaire
Travaux dirigés et pratiques
- TP 1 : installation de CUDA
- TP 2 : premiers programmes
- TP 3 : comparaison somme de vecteurs
- TP 4 : bioinformatique : parcimonie
- TP 5 : lancer de rayon
4. TP 4 : Bioinformatique : parcimonie
L'objectif du TP est de comparer les temps de calcul de différentes implantations de la fonction de Fitch qui calcule le maximum de parcimonie de deux séquences d'ADN.
On pourra s'inspirer du TP3 pour le programme à réaliser.
Dans l'exemple suivant, les données en entrée sont deux séquences x et y, et la séquence z est le résultat du calcul. On retourne le nombre de différences.
int parsimony(char *x, char *y, char *z, int size) {
int i, differences=0;
for (i = 0; i < size; i++) {
z[i] = x[i] & y[i];
if (z[i] == 0) {
z[i] = x[i] | y[i];
++differences;
}
}
return differences;
}
Le traitement à réaliser correspond à ce que l'on qualifie de réduction parallèle comme le produit scalaire.
4.1. Implantation sur le CPU
Ecrire une première version CPU de cette fonction et la tester.
4.2. Implantation sur le GPU - version 1
Ecrire une première version GPU de cette fonction et la tester :
- utiliser une grille 1D composée d'un seul bloc 1D de plusieurs threads
- stocker le nombre de changements dans la mémoire partagée (shared memory)
- réaliser la réduction sur la mémoire partagée
On rappelle que la mémoire partagée est locale à un bloc, tous les threads d'un même bloc peuvent y accéder.
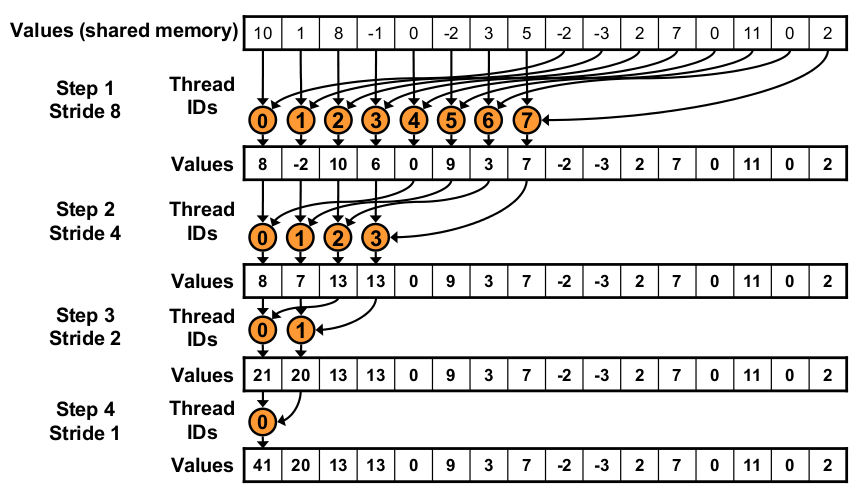
Tiré des transparents de Mark Harris (NVidia), on a ici un adressage dit séquentiel sans conflit d'accès aux bancs mémoire.
4.3. Implantation sur le GPU - version 2
Ecrire une seconde version GPU :
- utiliser une grille 1D composée de plusieurs blocs 1D de plusieurs threads
4.4. Implantation sur le GPU - version 3
On peut améliorer le traitement en évitant de coder le if, en tout cas cela fonctionne et apporte une amélioration sur le CPU :
char parsimony(char *x, char *y, char *z, int size) {
int i, differences=0;
for (i=0; i < size; ++i) {
unsigned char x_, y_;
int c;
x_ = x[i] & y[i];
y_ = x[i] | y[i];
z[i] = (x_ == 0) ? y_ : x_;
c = ((!x_) & 1);
differences += c;
}
return differences;
}