Ce site est en cours de reconstruction certains liens peuvent ne pas fonctionner ou certaines images peuvent ne pas s'afficher.
Cette page fait partie du cours de polytech PeiP1 et 2 Bio
11. Mise en pratique : Réseaux de Neurones
11.1. Introduction
Les Réseaux de Neurones ou Neural Networks sont une technologie ancienne dont les premiers travaux remontent à 1943 avec les travaux de McCulloch et Pitts.
Le réseau de neurones tente de modéliser le fonctionnement du cerveau au travers de neurones artificiels qui modélisent le fonctionnement de nos neurones cérébraux, ou tout du moins la vision que nous en avons.
Voici quelques références introductives concernant les réseaux de neurones :
- Réseaux de neurones
- What is a Neural Network ? (Vidéo)
- Introducing Deep Learning and Neural Networks for rookies
- Multi-Layer Neural Networks with Sigmoid Function
- Neural Networks Tutorial
11.2. Installation des packages
Pour que keras et tensorflow fonctionnent, il faut utiliser (Mai 2021) une version 3.6 de Python. Je conseille donc la procédure suivante sous Windows (et qui fonctionne également sous Linux) qui crée un environnement spécifique :
Dans le menu de Windows, chercher l'application Anaconda prompt et l'exécuter. Dans la console qui s'ouvre, taper les commandes suivantes et répondre 'y' à chaque fois que cela est demandé :
# Créer un environnement avec Python en version 3.6
# appelé nn pour Neural Network
(base)> conda create --name nn python=3.6
Vous devriez obtenir la sortie suivante :
Puis on installe les packages :
# rendre l'environnement nn actif
(base)> conda activate nn
# on vérifie qu'on a la bonne version de Python
(nn)> python --version
Python 3.6.13 :: Anaconda, Inc.
# on installe les packages requis pour l'environnement nn
(nn)> conda install numpy matplotlib seaborn pandas
(nn)> conda install scikit-learn
(nn)> conda install keras
Vous devriez avoir les packages sous les versions suivantes :
- keras 2.3.1
- matplotlib 3.3.4
- numpy 1.19.2
- pandas 1.1.3
- scipy 1.5.2
- seaborn 0.11.1
- tensorflow 2.1.0
Pour le vérifier, il suffit de saisir la commande conda list dans Anaconda prompt.
Enfin, pour pouvoir utiliser cet environnement avec Thonny, il faut modifier l'interpréteur comme sur l'image suivante (remplacez 'Jean-Michel' par votre nom utilisateur) :
Tout devrait fonctionner normalement. Si vous rencontrez un problème avec tensorflow / keras, essayez d'ajouter le code suivant en début de programme ce qui empêche l'utilisation de la carte graphique en cas de problème de compatibilité matérielle :
import os
os.environ['CUDA_VISIBLE_DEVICES'] = '-1'
11.3. Application
11.3.1. Les données
Nous allons créer un réseaux de neurones capable de prédire le diabète chez les indiens Pimas.
Les Pimas sont une une tribu d'Amérindiens vivant en Arizona et qui présente les plus forts taux d'obésité et de diabète de tous les États-Unis : record du monde du diabète de type 2 avec 50% de la population (contre 9% pour les blancs américains), âgée de plus de 35 ans. Certains attribuent cette obésité au fait que les Pimas n'ont plus la même alimentation qu'auparavant, pour d'autres il s'agit de facteurs génétiques.
Le fichier de données contient les informations suivantes pour chacune des 768 femmes de l'étude :
- Number of times pregnant (preg)
- Plasma glucose concentration a 2 hours in an oral glucose tolerance test (plas)
- for prediabetes : 40–199 mg/dL
- for diabete :200 mg/dL or greater
- Diastolic blood pressure (mm Hg) (pres)
- Triceps skin fold thickness (mm) (skin)
- 2-Hour serum insulin (mu U/ml) (insu)
- Body mass index (weight in kg/(height in m)^2) (bmi) indice de masse corporelle ou IMC : surpoids (25 à 30), obésité modérée (30 à 35), obésité sévère (35 à 40)
- Diabetes pedigree function (pedi)
- Age (years) (age)
- testé positif ou non au diabète (class)
11.3.2. Chargement des données et ajustements
On commence par charger les données avec pandas sous forme d'un DataFrame, puis on affiche les premières lignes du fichier :
preg plas pres skin insu bmi pedi age class
0 6 148 72 35 0 33.6 0.627 50 tested_positive
1 1 85 66 29 0 26.6 0.351 31 tested_negative
2 8 183 64 0 0 23.3 0.672 32 tested_positive
3 1 89 66 23 94 28.1 0.167 21 tested_negative
4 0 137 40 35 168 43.1 2.288 33 tested_positive
shape= (768, 9)
11.3.2.a Remplacement des données textes en données numériques
Toutes les données sont au format numérique sauf la dernière colonne, on va donc la remplacer par des valeurs numériques :
- tested_positive est remplacé par 1
- tested_negative est remplacé par 0
Warning: file_get_contents(polytech/rn_pimas_load_data.py): Failed to open stream: No such file or directory in /home/jeanmichel.richer/public_html/ez_web.php on line 397
Afficher le code polytech/rn_pimas_load_data.py
11.3.2.b Examen des données
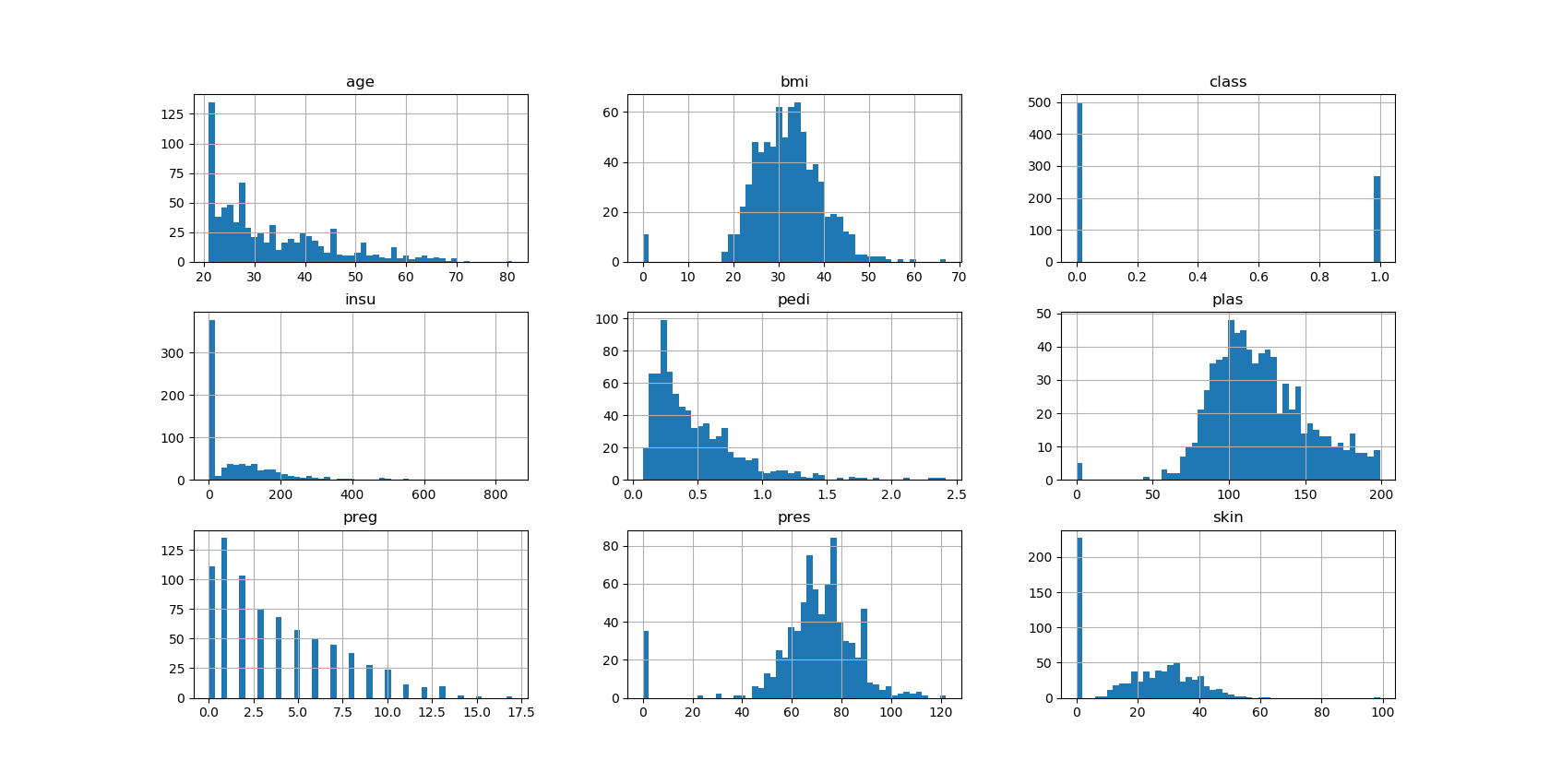
On examine ensuite les données en affichant un sommaire des données numériques ainsi qu'un graphique des distributions des données :
preg plas pres ... pedi age class
count 768.000000 768.000000 768.000000 ... 768.000000 768.000000 768.000000
mean 3.845052 120.894531 69.105469 ... 0.471876 33.240885 0.348958
std 3.369578 31.972618 19.355807 ... 0.331329 11.760232 0.476951
min 0.000000 0.000000 0.000000 ... 0.078000 21.000000 0.000000
25% 1.000000 99.000000 62.000000 ... 0.243750 24.000000 0.000000
50% 3.000000 117.000000 72.000000 ... 0.372500 29.000000 0.000000
75% 6.000000 140.250000 80.000000 ... 0.626250 41.000000 1.000000
max 17.000000 199.000000 122.000000 ... 2.420000 81.000000 1.000000
Par exemple, pour le nombre de grossesses, celui-ci varie de 0 à 17 !
Warning: file_get_contents(polytech/rn_pimas_examine.py): Failed to open stream: No such file or directory in /home/jeanmichel.richer/public_html/ez_web.php on line 397
Afficher le code polytech/rn_pimas_examine.py
11.3.2.c Traitement des données manquantes
Il apparaît que certaines données sont à 0, par exemple la pression artérielle (pres). Il s'agit probablement de données non connues qui par défaut apparaissent à 0. Dans ce cas, il est préférable de remplacer les valeurs nulles par la valeur médiane.
On note donc que les colonnes bmi, insu, plas, pres, skin possèdent des valeurs à 0 alors que ce ne devrait pas être le cas.
Warning: file_get_contents(polytech/rn_pimas_fill_missing.py): Failed to open stream: No such file or directory in /home/jeanmichel.richer/public_html/ez_web.php on line 397
Afficher le code polytech/rn_pimas_fill_missing.py
11.3.2.d Données d'apprentissage
Finalement, on crée les données d'apprentissage en séparant le DataFrame initial en la matrice des données d'apprentissage $X$ et la propriété à prédire $y$ :
- $X$ prend en compte les 8 propriétés suivantes : preg, plas, pres, skin, insu, bmi, pedi, age
- $y$ représente la présence ou non de diabète (class)
Warning: file_get_contents(polytech/rn_pimas_learning_data.py): Failed to open stream: No such file or directory in /home/jeanmichel.richer/public_html/ez_web.php on line 397
Afficher le code polytech/rn_pimas_learning_data.py
11.3.3. Création et apprentissage du réseau de neurones
A présent, la partie la plus sybilline commence.
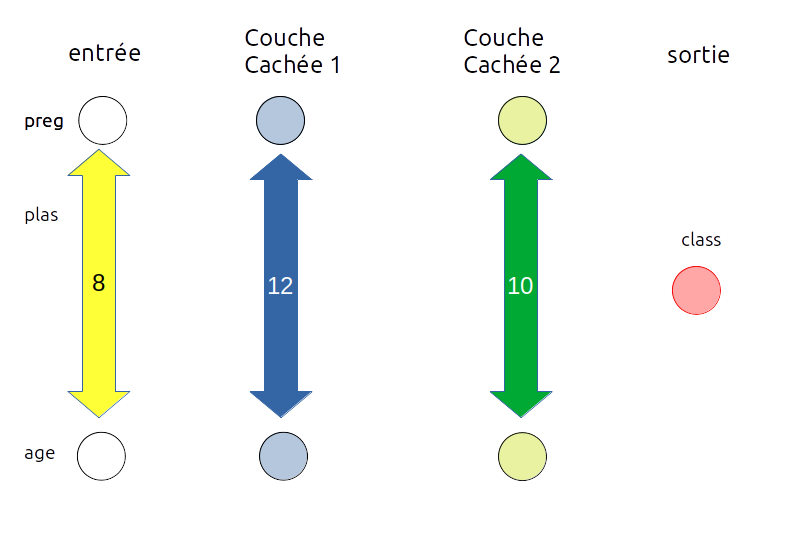
Nous utilisons Keras qui est une interface qui simplifie la création des RN. On met en place le réseau suivant :
- la couche d'entrée est composée de 8 neurones car chaque individu est caractérisé par 8 propriétés
- on crée ensuite deux couches cachées (hidden layers) :
- une première couche de 12 neurones
- une deuxième couche de 10 neurones
- la couche de sortie est composée d'un neurone qui correspond à la classe : est diabétique ou non
Le nombre de couches cachées et le nombre de neurones dans ces couches cachées est laissé au choix du concepteur du réseau de neurones.
Il est généralement nécessaire de faire plusieurs essais avec différentes couches
Warning: file_get_contents(polytech/rn_pimas_create_nn.py): Failed to open stream: No such file or directory in /home/jeanmichel.richer/public_html/ez_web.php on line 397
Afficher le code polytech/rn_pimas_create_nn.py
Warning: file_get_contents(polytech/rn_pimas_nn_learning.py): Failed to open stream: No such file or directory in /home/jeanmichel.richer/public_html/ez_web.php on line 397
Afficher le code polytech/rn_pimas_nn_learning.py
On définit trois paramètres (compile):
- la fonction a optimiser : binary_crossentropy
- l'optimiseur : adam
- les valeurs que l'on désire enregistrer et qui permettront de générer des graphiques : accuracy et mse (mean squared error)
Là, également, le choix est laissé libre à l'utilisateur de paramétrer son réseau :
- pour ce qui touche aux fonctions de perte (loss function), on pourra consulter ce site qui passe en revue les différentes fonctions : pour le problème qui nous intéresse, il est recommandé d'utiliser la fonction Binary Cross-Entropy, puis pour la couche de sortie, une sigmoïde.
- concernant les optimiseurs, on a le choix entre :
- Adam, Adagram, Adadelta, Adamax
- SGD (Stochastic Gradient Descent)
- RMSProp
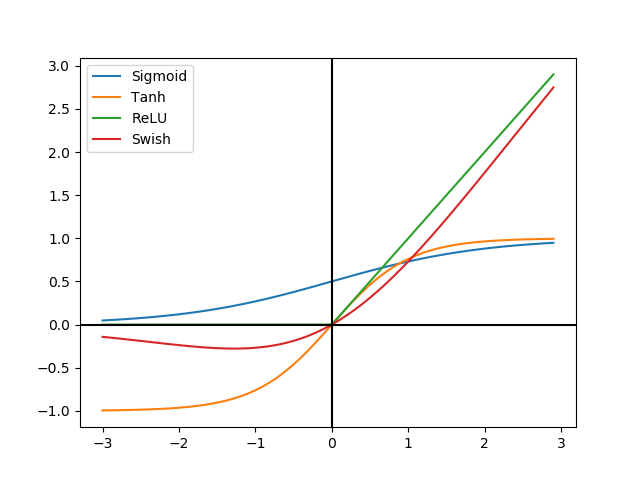
- pour les fonctions d'activation :
- linear, elu (Exponential linear Unit), relu (Rectified Linear Unit), selu (Scaled Exponential Linear Unit)
- softmax, softplus, softsign
- exponential, tanh (tangente hyperbolique), sigmoid
- pour le nombre d'epoch, il faut choisir une valeur entre 500 et 5000 dans le cas présent.
11.3.4. Résultat et précision du RN
On récupère les informations liées à l'apprentissage du RN (evaluate) et on les affiche :
Warning: file_get_contents(polytech/rn_pimas_precision.py): Failed to open stream: No such file or directory in /home/jeanmichel.richer/public_html/ez_web.php on line 397
Afficher le code polytech/rn_pimas_precision.py
# pour 8000 epochs
loss = 0.0335
accuracy = 98.6979 %
mse = 0.0094Les graphiques qui correspondent aux données enregistrées (mse, accuracy) sont les suivants :
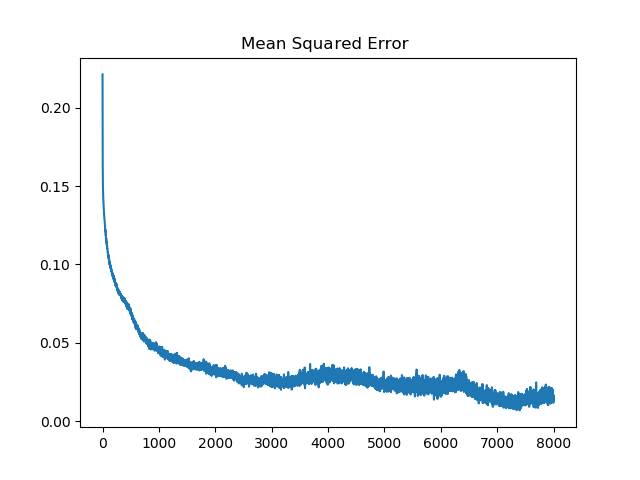
Progression Mean Squared Error (8000 epochs)
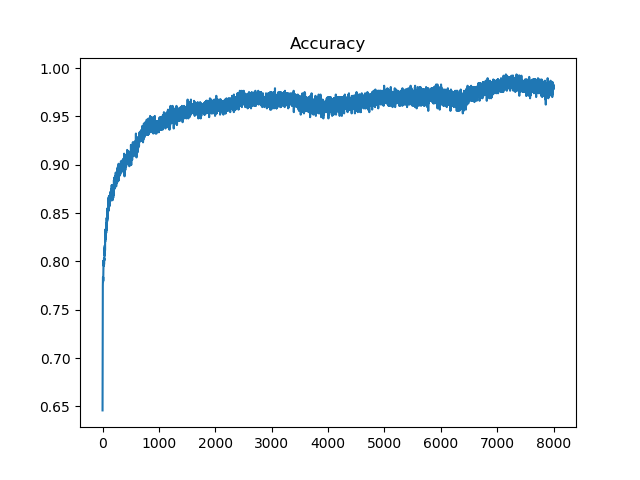
Progression précision (Accuracy, 8000 epochs)
On remarque que $mse ≈ 1 / { accuracy}$, i.e. plus l'erreur est faible, meilleure est la précision.
Enfin, on essaye de réaliser la prédiction sur $X$ à partir du RN et on compare à ce qu'on devrait avoir, c'est à dire $y$ :
Warning: file_get_contents(polytech/rn_pimas_prediction.py): Failed to open stream: No such file or directory in /home/jeanmichel.richer/public_html/ez_web.php on line 397
Afficher le code polytech/rn_pimas_prediction.py
expected=1, found=0
expected=0, found=1
expected=0, found=1
expected=1, found=0
expected=0, found=1
expected=1, found=0
expected=0, found=1
expected=1, found=0
expected=0, found=1
expected=1, found=0
Exercice 11.1
A partir de ce qui a été vu, créer un seul fichier nommé rn_pimas.py afin de réaliser la prédiction du diabète chez les Pimas.
Quel est la précision du RN pour 1000 epochs ?
11.3.5. Amélioration de la prédiction
Peut-on améliorer la prédiction ?
11.3.5.a Normalisation / Standardisation
La plupart des algorithmes d'apprentissage apprennent mieux si les données sont normalisées ou standardisées.
Le fait que les données possèdent des échelles différentes (ex: age de 0 à 80, nombre de grossesses variant de 0 à 17) semble en effet influer sur la capacité à apprendre.
On utilise donc une mise à l'échelle basée sur les valeurs minimales et maximales d'une propriété (cf. sklearn.preprocessing).
Warning: file_get_contents(polytech/rn_pimas_normalization.py): Failed to open stream: No such file or directory in /home/jeanmichel.richer/public_html/ez_web.php on line 397
Afficher le code polytech/rn_pimas_normalization.py
On place donc la normalisation des données avant la création du réseau de neurones.
Exercice 11.2
Reprendre le fichier rn_pimas.py et ajouter la normalisation des données.
Quel est alors la précision du RN avec 1000 epochs ?
11.3.5.b Batch_size
Le paramètre epoch correspond à une phase d'apprentissage : on propage les calculs à travers le RN puis on effectue la rétro-propagation, c'est à dire la mise à jour des poids des associés aux neurones.
Le paramètre batch_size définit le nombre d'individus utilisés pour une phase d'apprentissage.
Sur un AMD Ryzen 5 3600 doté d'une carte graphique GTX 1070 les temps de calcul pour 8000 epochs en fonction de la variable batch_size sont de :
- GPU :
- batch_size = 16 : 664 s (accuracy = 94.5312)
- batch_size = 32 : 341 s (accuracy = 99.7396)
- batch_size = 64 : 174 s (accuracy = 95.0521)
- batch_size = 128 : 92 s (accuracy = 97.3958)
- batch_size = 256 : 50 s (accuracy = 95.0521)
- batch_size = 384 : 37 s (accuracy = 93.4896)
- batch_size = 768 : 23 s (accuracy = 93.4896)
- CPU :
- batch_size = 5 : 868 s (accuracy = 89.9740)
- batch_size = 8 : 549 s (accuracy = 92.4479)
- batch_size = 10 : 440 s (accuracy = 95.7031)
- batch_size = 16 : 279 s (accuracy = 97.2656)
- batch_size = 32 : 147 s (accuracy = 99.7396)
- batch_size = 64 : 77 s (accuracy = 98.1771)
- batch_size = 128 : 40 s (accuracy = 96.8750)
- batch_size = 256 : 23 s (accuracy = 94.7917)
- batch_size = 384 : 18 s (accuracy = 94.0104)
- batch_size = 768 : 12 s (accuracy = 94.6615)
Dans le cas présent, on obtient une meilleure prédiction avec un batch_size = 32.
Exercice 11.3
Reprendre le fichier rn_pimas.py et modifier le batch_size.
Quelle est alors la précision du Réseau de Neurones avec 1000 epochs ?
Exercice 11.4
Analyser le fichier des indiens Pimas en créant un classifieur SVM.
Quelle est la précision obtenue et avec quels paramètres ?
Comparer les résultats avec un réseau de neurones.